GUÍA BÁSICA DE PRE-PROCESAMIENTO MEDIANTE EEGLAB
Autores:
- Carolina Serna, email: carolinasernarojas@gmail.com
- Francisco García, email: fpretelt@gmail.com
Laboratorio de neurofisiología.
Introducción a EEGLAB
EEGLAB es una colección de funciones de MATLAB que pueden ser llamadas desde una interfaz gráfica principal o a través de comandos de línea [1]. En esta guía se expondrán diferentes pasos a seguir para pre-procesar los registros de las dos maneras; sin embargo es una información básica que puede ser ampliada en las referencias mencionadas para cada sección.
El mecanismo de historia de EEGLAB EEG.history guarda todas las operaciones que se han ejecutado en los datasets desde la interfaz gráfica, lo que permite conocer qué comandos se han utilizado para determinados procesos realizados y construir líneas de código que puedan hacer en gran medida un procesamiento automático de múltiples señales. De este modo, es importante conocer la estructura de los datos EEG y las subestructuras principales de éstos, como son: EEG.data, EEG.event, EEG.urevent, EEG.epoch, EEG.chanlocs and EEG.history [1].
EEGLAB utiliza cinco nombres de variables reservadas para su uso :
- EEG - Actual dataset EEG
- ALLEEG - Colección de todos los datasets EEG cargados
- CURRENTSET - El índice de dataset actuales
- LASTCOM - El último comando utilizado desde el menú EEGLAB
- ALLCOM - Todos los comandos utilizados desde el menú EEGLAB
Se puede acceder a cada una de ellas o a las subestructuras escribiendo en la ventana de comandos de MATLAB el nombre de la variable que desea ser vista [1], por ejemplo:
>> ALLEEG(1)
Retorna la estructura del primer dataset cargado en el workspace EEGLAB/MATLAB con toda la información de éste [1].
>> EEG
Incluye el nombre del archivo, el número y la locación de los canales, la frecuencia de muestreo de los datos, el número de épocas o ensayos, información acerca de los eventos, cada una de las épocas, entre otros [2]. Para acceder a un dataset que se ha importado o guardado en ese momento y se desea ver en la interfaz gráfica, se utiliza el siguiente comando » eeglab redraw. Si hay abierta una sesión de EEGLAB primero escriba >[ALLEEG EEG CURRENTSET] = eeg_store(ALLEEG, EEG) para guardar la información modificada y luego el comando anterior [1].
Estructuras de datos de EEGLAB
Sección utilizada para el uso de EEGLAB y el uso de las funciones en los scripts de MATLAB [1].
La estructura EEG contiene toda la información del dataset actual cargado a EEGLAB. Para ver lo que incluye esta estructura y lo que se ha procesado en el dataset, se puede proceder en la ventana de comandos de MATLAB con » EEG y aparecerán las siguientes líneas de comandos (ver figura 1) [2].

EEGLAB pop_functions
Las funciones con el prefijo pop_ o eeg_ son funciones que toman la estructura EEG como su primer argumento.Las funciones con el prefijo pop_ pueden ser llamadas ya sea desde el menú de EEGLAB o por un comando de línea, mientras que las funciones con el prefijo eeg_ sólo pueden ser llamadas con un comando de línea de MATLAB. Las funciones pop_ pueden ser llamadas desde el menú de EEGLAB a través de las diferentes secciones y abrirán una ventana para llenar una serie de parámetros y continuar con el procesamiento. Luego aparecerá en el historial de EEG las funciones con los parámetros utilizados para ser generadas desde unas líneas de comandos. Si la función es llamada sólo con el primer parámetro EEG, se abrirá una ventana de consulta interactiva para ser llenada; pero si no se remueven o cambian esos parámetros, el procesamiento se hará directamente (sin abrir ninguna ventana) con las indicaciones dadas anteriormente [1].
Por ejemplo, si se escribe >> EEG = pop_eegfilt(EEG) en la ventana de comandos, esto abrirá una ventana interactiva permitiendo filtrar los datos de acuerdo a unos parámetros de entrada. Si se desea filtrar el dataset EEG sin una interfaz gráfica se puede escribir por ejemplo » EEG = pop_eegfilt( EEG, 1, 0), lo cual es un filtro pasa altas que filtrará los datos por encima de 1 Hz [1].
Para ver el estado del dataset actual utilice la función >> EEG = eeg_checkset(EEG). Ésta también sirve para revisar la estructura del evento y es llamada cada que el dataset es modificado, convirtiendo valores numéricos a cadenas si es necesario [1,3].
Cargar datos a EEGLAB
Instalación de EEGLAB:
Primero es necesario descargar la toolbox EEGLAB en la siguiente página https://sccn.ucsd.edu/eeglab/downloadtoolbox.php. Una vez descomprimido el archivo EEGLAB, obtendrá una carpeta nombrada “eeglabxxxx” (dependiendo de la versión descargada) [3].
Inicie MATLAB y agregue la carpeta “eeglabxxxx” al path de MATLAB de la siguiente manera (ver figura 2) [3]:
Home(file) > Set path > Add folder > ok > Save
En el menú home (o file dependiendo de la versión) seleccione Set Path, luego la carpeta “eeglabxxxx” y para finalizar Save. Esto permitirá a la toolbox EEGLAB estar disponible para futuras sesiones de MATLAB. Tenga en cuenta que si ya presenta alguna versión previa de EEGLAB en el path de MATLAB, es recomendable desinstalarla antes de proceder con la nueva (seleccione la versión que desea retirar, luego elija Remove) [3].
Si desea agregar la toolbox de EEGLAB a MATLAB mediante código, ingrese una de las siguientes líneas en la ventana de comandos de MATLAB [3]:
>> pathtool
La cual abrirá una ventana como la mostrada en la figura 2, en la cual deberá seguir los pasos indicados anteriormente.
>> addpath(genpath('dirección'));
Donde dirección representa la ubicación actual de la carpeta ‘eeglabxxxx’. Esta opción agrega la carpeta al path, sin embargo no la guarda, por lo que será necesario ejecutar la línea cada vez que abra MATLAB.
Iniciando EEGLAB:
Para iniciar EEGLAB, ejecute la siguiente línea en la ventana de comandos de MATLAB [3]:
>> eeglab;
O para iniciar EEGLAB desde comandos de línea e inicializar varias variables » [ALLEEG EEG CURRENTSET ALLCOM] = eeglab [1,3].
Una vez cargada la toolbox se mostrará la ventana principal de EEGLAB (ver figura 3) [3].
En la sección superior, encontrará típicamente los menús File, Edit, Tools, Plot, Study, Datasets, Help [3].
Abrir un dataset ya existente:
Para cargar un dataset ya existente, es decir, en un formato reconocible por EEGLAB (generalmente .set), se debe seleccionar File > Load existing dataset y se mostrará una ventana similar a la figura 4 [3].
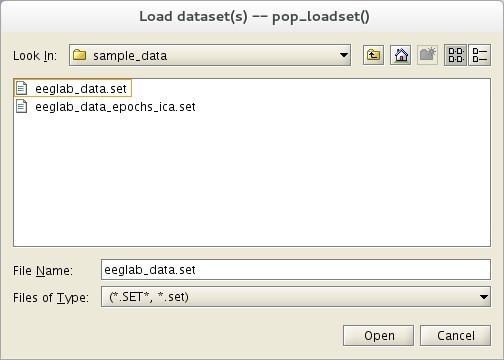
Si desea cargar un dataset ya existente mediante código, ejecute la siguiente línea en la ventana de comandos de MATLAB [1]:
>> EEG=pop_loadset;
La cual abrirá una ventana similar a la figura 4 y deberá elegir el dataset a cargar. Por otro lado, si desea cargar un dataset del cual conoce la ubicación y nombre, puede hacerlo directamente ejecutando el siguiente código [1]:
>> EEG = pop_loadset('filename','filedir');
Donde filename representa el nombre del dataset a cargar (incluir la extensión, por ejemplo .set) y filedir representa la dirección de la carpeta donde se encuentra el archivo. Recuerde ingresar los campos filename y filedir entre comillas simples (‘’) y en la variable EEG quedará registrada la información del dataset seleccionado.
**Importación de datasets:**
EEGLAB reconoce como propias algunas extensiones como .set y .fdt, sin embargo, es usual encontrar diversos formatos que requieren ser importados antes de poder procesar este tipo de datos con EEGLAB; ejemplo de estos son EDF (European Data Format), EDF+ (European Data Format Plus), CNT (Neuroscan file) y BDF (Biosemi file); los datos también pueden ser importados directamente desde un arreglo de MATLAB.
Si desea importar datasets desde la interfaz gráfica de EEGLAB, ir al menú File > Import data > Using EEGLAB functions and plugins. EEGLAB ofrece una gama de opciones para importar distintos formatos, elija el formato correspondiente y siga las instrucciones (los plugins y funciones de MATLAB asociadas a importar distintos formatos pueden variar de versión a versión). La importación de datos puede ser un proceso inestable dependiendo de la versión y el formato a importar, por tanto se recomienda verificar los datos una vez importados [4].
● Importación desde un arreglo de MATLAB: La variable debe ser un arreglo en 2D, representando cada una de las filas un canal y cada una de las columnas un punto distinto (datos). Del menú File > Import data > Using EEGLAB functions and plugins seleccione from ascii/float file or Matlab array y se mostrará una ventana similar a la figura 5 [4].
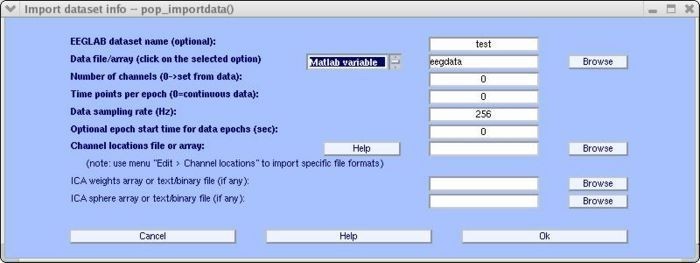
Además de importar datos mediante un arreglo de MATLAB, es posible importar datos desde un archivo .mat o mediante un archivo de texto ASCII seleccionando la opción respectiva en el apartado Data file/array, indicando en la caja de texto el nombre de la variable o archivo (en caso de ser un archivo, éste debe encontrarse en el directorio actual de MATLAB). Las casillas Number of channels, Time points per epoch y optional epoch start time for data epochs suelen ser dejadas por defecto y en el campo Data sampling rate se indica la frecuencia de muestreo en Hz [4]. Finalmente, indicar el archivo que contiene la ubicación de los canales en el apartado Channel locations file or array (es opcional, este archivo puede ser añadido posteriormente). En próximas secciones se tratará la importancia de los archivos asociados a la ubicación de los canales y su manejo mediante EEGLAB[4].
Después de rellenar los campos deseados y presionar Ok se mostrará una ventana similar a la figura 6, (completar el campo File to save dataset en caso de querer guardar el dataset en una dirección) y presionar de nuevo Ok [4].
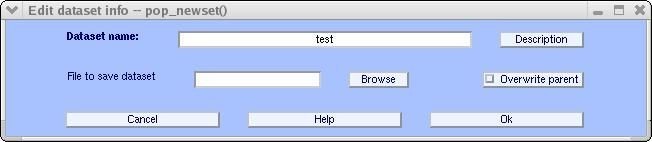
● Importación desde archivo .EDF: Para importar datasets desde EDF de 16
bits, ir al menu File > Import data > Using EEGLAB functions and plugins > From EDF/EDF+ Files y seleccionar el dataset a importar. Luego de importarlo se mostrará una ventana parecida a la figura 6, rellene los campos deseados y presione Ok [4]. La importación desde archivos EDF puede, en algunos casos, presentar problemas o inconsistencias; si la importación desde la interfaz gráfica de EEGLAB no es exitosa, lea la “Guía de importación EDF” del Laboratorio de neurofisiología.
Si se desea extraer eventos desde un archivo EDF, ir al menú File > Import event info > From data channel [4].
● Importación desde archivo .CNT: Para importar datasets desde CNT ir al menú File > Import data > From .CNT data file y seleccione el archivo a convertir. Luego de seleccionar el archivo CNT se mostrará una ventana parecida a la figura 7 [4].
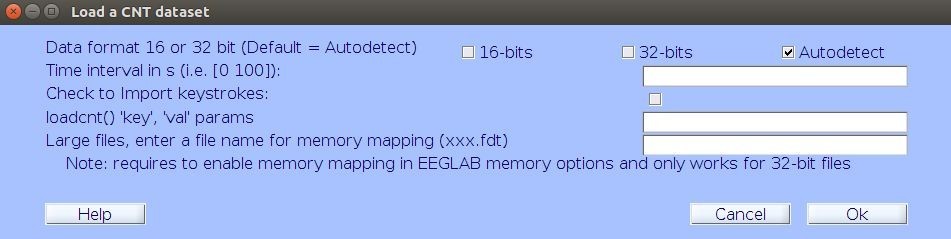
Presione Ok y posteriormente será mostrada una ventana similar a la figura 6; después de rellenar los campos deseados presione Ok nuevamente [4]. Si desea importar archivos CNT mediante script, lea la “Guía de preprocesamiento PREP” del Laboratorio de neurofisiología.
Importación, adición y modificación de eventos
La subestructura EEG.event contiene los registros de los eventos experimentales que ocurrieron mientras se hizo el registro EEG. Ésta guarda en general tres campos: type, latency y urevent (ver figura 8) [2].

-
type tipo de evento
-
latency latencia del evento
-
urevent índice de los eventos originales.
También puede contener duration, campo reconocido por EEGLAB que guarda la información acerca de la duración del evento; se muestra en segundos para datos continuos y en milisegundos para datos con épocas. El campo position observado en la figura 8 cumple propósitos informativos y puede ser modificado por el usuario para que indique información relevante de cualquier naturaleza y no solo posición[2,5].
Campos como epoch, duration, urevent, son creados automáticamente durante la extracción de épocas, rechazo de datos o almacenamiento de eventos [5].
EEG.urevent guarda toda la información que fue originalmente cargada dentro del dataset más los eventos que fueron añadidos manualmente por el usuario. Cuando se remueven eventos del dataset por el rechazo de artefactos o extracción de épocas, los eventos originales continúan existiendo sólo en la estructura urevent[2,5].
El campo epoch contiene los índices de las épocas en los datos (si las hay) y los eventos que hay dentro de éstas [5].
Nota:
-
Los campos latency y duration no están registrados en segundos, están registrados en puntos por señal, es decir, el número de muestras que han transcurrido desde el inicio del registro hasta el evento, y EEGLAB lo reconoce de esta manera al trabajar mediante script. Los datos ingresados mediante interfaz gráfica suelen ser en segundos [2,5].
-
En continuos datasets, EEGLAB puede añadir eventos tipo ‘boundary’ para especificar límites en los datos, es decir, una discontinuidad dentro de los datos continuos. Estos ‘boundary’ también pueden ser creados automáticamente cuando porciones de los datos continuos son rechazados o cuando datasets continuos son concatenados; indican la latencia y a veces la duración de esas discontinuidades que estas operaciones introducen en los datos [5].
-
Todos los campos pueden contener números (1, 2, etc.) y cadenas (por ejemplo ‘square’, ‘rt’, etc.) pero no mixtos, excepto para latency, duration, urevent y epoch que deben contener sólo números [5].
Los campos type y latency son los campos más importantes para los eventos en EEGLAB, ya que algunas funciones reconocen y usan estos dos campos para graficar los eventos, hacer pruebas de clasificación, etc [5].
Para ver información de los eventos escriba en la ventana de comandos de MATLAB:
>> EEG.event.nombredelarchivo
Por ejemplo para ver la latencia de los eventos:
>> {EEG.event(1:5).latency}
El cual retorna las primeras 5 latencias del dataset : [129.0087] [218.0087] [267.5481] [603.0087] [659.9726]
Para ver estas latencias en segundos en lugar de puntos de muestreo de la señal, primero se necesita convertir esta matriz de celda a una matriz numérica ordinaria usando la función de MATLAB cell2mat. Luego restar 1 porque el primer punto de la muestra corresponde al tiempo 0 y finalmente dividir por la frecuencia de muestreo,así [5] :
>>(cell2mat({EEG.event(1:5).latency})-1)/EEG.srate
Lo cual devuelve las mismas latencias de los eventos en segundos : ans = 1.0001 1.6954 2.0824 4.7032 5.1482
**Importación y adición de eventos:**
Los eventos pueden ser importados en diferentes formatos dentro de EEGLAB, entre los que se encuentran: importación desde un canal, un arreglo en MATLAB o un archivo ascii. Solo hay que seleccionar en el menú de la interfaz gráfica File > Import event info. Este mismo método se puede usar para añadir nuevos eventos a una tabla preexistente [6].
Importación de eventos desde un canal: Es usual que los datos asociados a eventos estén codificados en una de las filas de la matriz EEG (canales). En un dataset de 33 canales, el último corresponde a la información de eventos (en caso de presentar este tipo de almacenamiento, por ejemplo en formatos EDF), y contendrá 1 (inicio de estímulo), 2 (respuesta del sujeto) o 0 (otros). Para importar eventos desde un canal, ir al menú File > Import event info > from data channel y se activará una ventana como la mostrada en la figura 9 [6].
En el campo Event channel(s) indicar el canal asociado a los eventos que es usualmente el último y colocar el puntero sobre los otros campos para más información. Además si desea que el canal asociado a los eventos (el cual no contiene datos asociados a voltajes) sea eliminado luego de la importación, active la casilla respectiva [6].
Importación de eventos desde un arreglo de MATLAB o archivo de texto (ascii: La importación de eventos también es posible desde un arreglo de MATLAB o un archivo de texto (ascii). La estructura del archivo se ilustra en la tabla 1,note que la latencia está en segundos [6].
Tabla 1. Formato de importación de eventos desde archivo de texto (ascii) o arreglo en MATLAB [6].
Si se desea importar eventos desde un archivo de texto o arreglo en MATLAB ir al menú File > Import event info > Import Matlab array or ASCII file; se activará una ventana como la vista en la figura 10 [6].
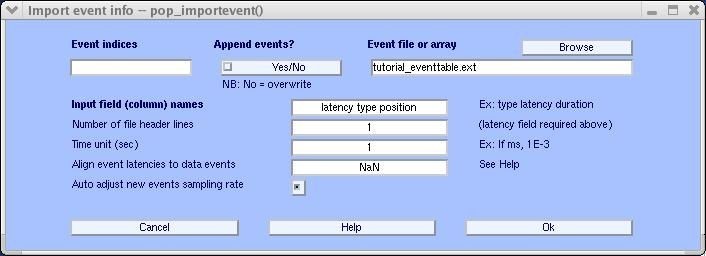
En el campo Input field names indicar los nombres asociados a las columnas en el arreglo (en el caso de la tabla 1: latency, type y position). Colocar 1 en el campo Number of file header lines, ya que la primera fila da el nombre de las columnas. Las columnas asociadas a latency y type son necesarias para la importación, mientras que el campo position puede ser modificado por el usuario (es necesario completar los campos en minúscula, debido a que MATLAB es sensible a mayúsculas). Los campos Event indices y Append events? son usados para reemplazar eventos antiguos por nuevos o para agregar los eventos importados a una lista de eventos existentes,respectivamente [6].
Nota: En muchos casos como se mencionó anteriormente, información importante relacionada a los eventos se guarda en un canal extra al momento de realizar el registro; sin embargo, información más detallada es guardada en un archivo de texto. Si se desea sincronizar la información existente en un dataset con una codificada en un archivo de texto, es necesario implementar el campo Align event latencies to data events, en el cual 0 significa que el primer evento del archivo de texto corresponde al primer evento del dataset, 1 significa que el segundo evento en el archivo de texto corresponde al segundo evento en el dataset y así sucesivamente. La opción NaN indica que esta opción será ignorada y también es válido ingresar números negativos indicando que los eventos asociados al archivo de texto iniciaron antes que los registrados en el dataset. La última casilla auto adjust new events sampling rate es usada para ajustar la frecuencia de muestreo asociada a los eventos en el archivo con la frecuencia indicada en el dataset, sin embargo, esto puede provocar ligeros desfases temporales que en registros largos podrían ser de importancia [6].
Insertar eventos manualmente: Para insertar nuevos eventos manualmente seleccione en el menú Edit > Event values. Haga clic en el botón Insert event para agregar un nuevo evento antes del evento actual y clic en Append event para añadir uno después del evento actual (ver figura 11). Luego se mostrará la información del evento para ser modificada, por ejemplo para insertar un evento tipo new modifique el campo type con new y 500 en latency, haga clic en Ok o la información no será guardada. Además, hay que considerar que los eventos se presentan en el orden en el que aumenta la latencia [5].
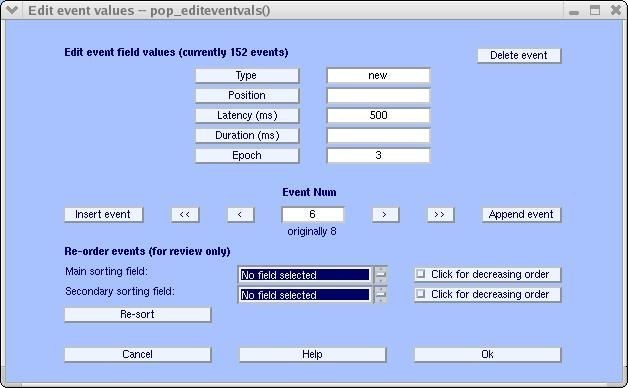
Otra manera de insertar eventos nuevos es seleccionando el menú Edit > Event fields [5]:
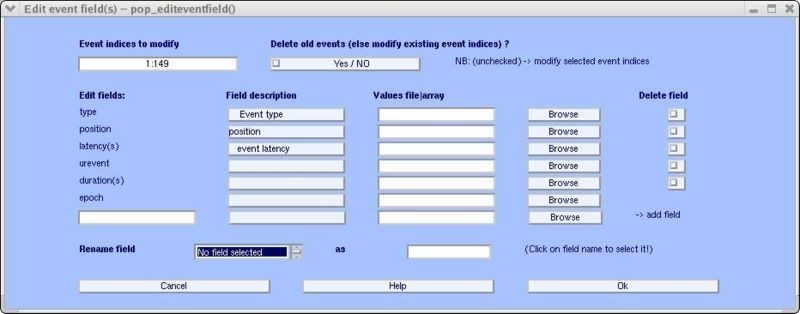
En esta interfaz gráfica (figura 12) se pueden añadir nuevos campos e importar matrices de MATLAB o archivos de texto; eliminar campos o renombrarlos y cambiar también la descripción de los campos dando clic en los botones de la columna Field description. Cuando haya terminado de editar presione Ok [5].
Importación de archivos de información E-Prime: El formato E-Prime es altamente configurable. Es posible importar este tipo de datos al igual que se importa un archivo de texto o un arreglo en MATLAB, configurando las columnas con los nombres indicados en el archivo E-Prime; sin embargo, es posible que en algunos casos sea necesario exportar los datos E-Prime a archivos delimitados por celdas (como hojas de cálculo) para editar información de las columnas que podría no ser leída correctamente por MATLAB [6].
Importación de archivos de información .DAT (neuroscan): Para importar el archivo .DAT asociado a un archivo .CNT cargado previamente, ir al menú File > Import epoch info > From Neuroscan .DAT info file. Luego de seleccionar el archivo deseado, emergerá una ventana como la mostrada en la figura 13 [6].
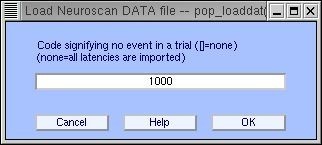
En la importación de archivos .DAT, cada época debe contener un tiempo de reacción en milisegundos; aunque es posible que alguna época no tenga tiempo de reacción (si el sujeto no responde, por ejemplo). En estos casos se recomienda establecer un valor código, que indique la ausencia de tiempo de respuesta, por ejemplo, un tiempo de reacción de 1000 ms indicará que el tiempo de respuesta es nulo, pero si todas las épocas del procedimiento tienen ya asociado un tiempo de reacción, no ingrese valor alguno en este paso [6].
Edición de eventos:
Para editar valores de los eventos seleccione el menú Edit > Event values. Se mostrará una ventana similar a la figura 14 [3].
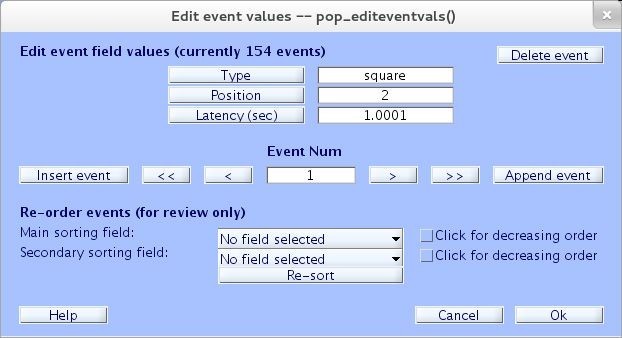
El campo type hace referencia al tipo de evento, es información usada para diferenciar entre distintos grupos de eventos que puedan existir en un estudio (por ejemplo, aciertos – errores). El segundo campo (Position , el nombre de este campo puede variar con las necesidades del usuario) es puramente informativo y en él puede ser almacenada cualquier información de interés; en el ejemplo de la figura 14, hace referencia a la ubicación en pantalla del estímulo mostrado en el paradigma. Finalmente, el campo Latency corresponde al instante de tiempo en segundos en el que fue registrado el evento [3].
La información de los eventos se guarda en la subestructura event dentro de la estructura EEG (o cualquier otra estructura asociada a algún registro), de manera que si se desea modificar la información de un evento mediante código, basta con modificar la subestructura event.
En la interfaz gráfica mostrada en la figura 14 (eventos actuales 154), se encuentran todos los eventos experimentales del registro que pueden ser modificados simplemente escribiendo los nuevos valores en el espacio de edición para cada campo. También pueden ser eliminados pulsando en el botón Delete event [5].
El botón Re-sort reordena los eventos y es solo por fines de visualización, ya que EEGLAB no puede procesar una estructura de evento que no esté ordenada por el incrementos de la latencia del evento [5].
Visualización de datos (Scroll):
Para visualizar el scroll de datos ir al menú Plot > Scroll data. A continuación se visualizará una ventana como la ilustrada por la figura 15 [3].
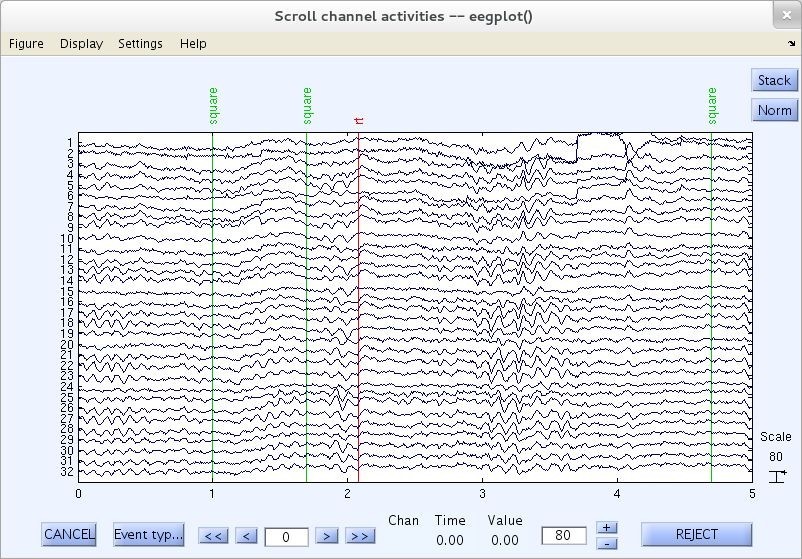
El eje x de las gráficas representa el tiempo en segundos mientras el eje y representa el voltaje, cuya escala en microvoltios se encuentra en la esquina derecha y puede ser modificada en la caja de texto ubicada en la parte inferior derecha de la ventana (en la figura 15 la escala visualizada es de 80 microvoltios)[4].
Para cambiar el tiempo mostrado en la ventana del scroll ir al menú Settings > Time range to display. En la ventana emergente ingrese el valor en segundos a mostrar. En la figura 16 se evidencia el efecto de cambiar el intervalo temporal de los 5 segundos originales (figura 15) a 10 segundos [3].
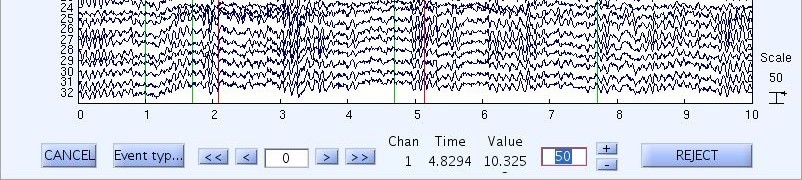
Cada una de las líneas curvas mostradas en el scroll corresponde a un canal del registro, el número asociado a cada canal se visualiza a la izquierda de la ventana. Es posible cambiar la cantidad de canales visualizados mediante el menú Settings > Number of channels to display [3].
Para acercar una sección en específico del scroll, ir al menu Settings > Zoom off/on > Zoom on y seleccionar el área de interés usando el mouse. Presionar clic derecho para deshacer el acercamiento y para salir de la opción zoom ir al menú Settings > Zoom off/on > Zoom off [3].
Si se busca activar líneas guía tanto para el eje x como para el eje y, ir al menú Display > Grid > X grid on o Display > Grid > Y grid on respectivamente. Repetir el proceso para desactivar las líneas (X grid off o Y grid off) [3].
Si se desea observar el scroll de un dataset mediante código, ingresar en la ventana de comandos de MATLAB [1]:
>> pop_eegplot( EEG, 1, 1, 1); donde EEG es la estructura asociada al dataset de interés [1].
● Rechazar datos visualmente: Se realiza con el fin de eliminar segmentos del registro que presenten un movimiento brusco del paciente o señales planas por desajuste del caso. Estos segmentos pueden afectar el preprocesamiento posterior, también pueden marcar interpolación de más electrodos en el caso de preprocesamiento PREP o afectar la implementación de la técnica de ICA[3].
La inspección se realiza por medio de la interfaz de EEGLAB y es recomendable en los registros realizados en descanso. En los registros realizados bajo una tarea se debe tener precaución con las marcas y considerar la ventana de tiempo a analizar (época) [3].
Si se desea eliminar datos mediante inspección visual, ir al menú Plot > Scroll data o al menú Tools > Reject continuous data. Seleccionar la sección a eliminar y pulsar el botón Reject; es posible marcar más de una región con el fin de eliminarla. Además, para desmarcar una región seleccionada, solo hay que hacer clic una vez sobre ésta. En la figura 17 se visualizan regiones marcadas para su eliminación [3].
Nota: para marcar una región es necesario que esté desactivada la opción de zoom.
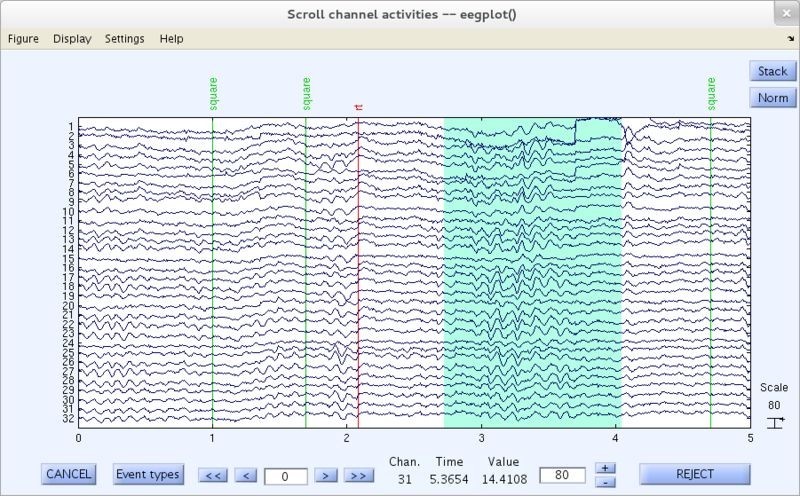
Luego de pulsar Reject, una ventana emergente pedirá la información básica del nuevo dataset (sin las regiones eliminadas), como nombre y dirección en caso de querer guardar el registro, una vez indicada la información pulsar Ok [3].
Recuerde que el rechazo visual de épocas se hace desde la ventana del scroll, por lo que si se quiere implementar la inspección visual de épocas en el código basta con ejecutar:
>> pop_eegplot( EEG, 1, 1, 1);
Y seguir las instrucciones dadas anteriormente. EEG es el nombre de la estructura asociada al dataset en cuestión [3].
● Eliminar dataset de la memoria RAM: Debido a la cantidad de datos involucrada en ciertas ocasiones es de utilidad eliminar del espacio de trabajo de MATLAB datasets que no se necesiten inminentemente (se trata de eliminarlos de la memoria RAM, no se modificará el archivo almacenado en el disco duro, en caso de haber guardado los datos). Para eliminar de la memoria determinado dataset, ir al menú File > Clear dataset(s) o Edit Delete dataset(s) y en la ventana emergente, indicar el índice (o los índices,separados por un espacio) numérico del dataset a eliminar. Si desea eliminar un dataset mediante script ejecute la siguiente línea en la ventana de comandos [3]:
>> clear EEG;
Donde EEG es la estructura asociada al dataset en cuestión.
Ubicación de los canales
La ubicación de los electrodos (canales) al momento de realizar el registro no es aleatoria, viene dada por distribuciones estándar pensadas para promover la reproducibilidad en registros EEG y optimizar la captación de información [7].
Configuración internacional 10-20**: </></u>
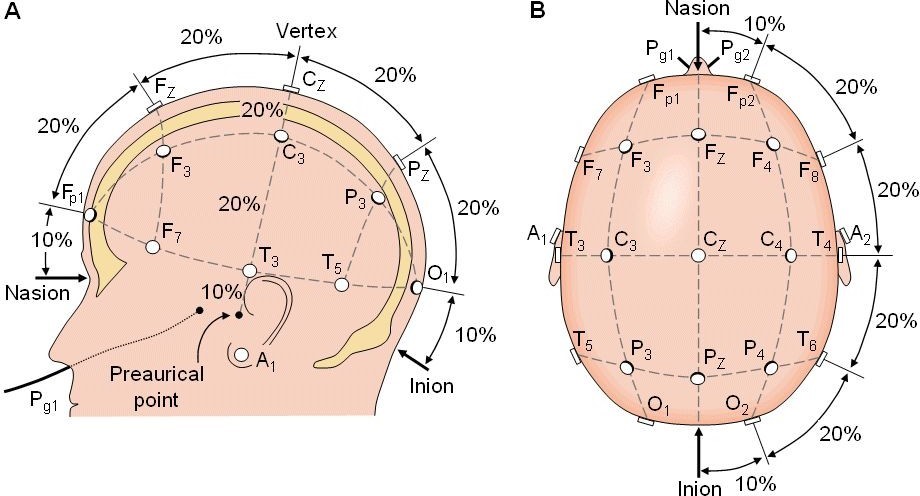
La configuración internacional 10-20 (figura 18) se mantuvo durante medio siglo como la configuración estándar para la ubicación de electrodos en electroencefalografía. Este sistema, describe la posición de los sensores en la superficie de la cabeza mediante la distancia relativa entre puntos de referencia en la superficie craneal. El objetivo original del sistema, era proveer una metodología reproducible para colocar un número de electrodos de EEG relativamente bajo, generalmente 21 [7].
Configuración internacional 10-10
Con la necesidad de una mayor densidad de electrodos, se creó el sistema internacional 10-10 (figura 19), también conocido como el sistema 10% o el sistema 10-20 extendido, pudiendo ubicar con él hasta 81 electrodos, sin embargo, en la actualidad existen cascos comerciales con mayor densidad de sensores, que van desde 128 hasta 256 electrodos.
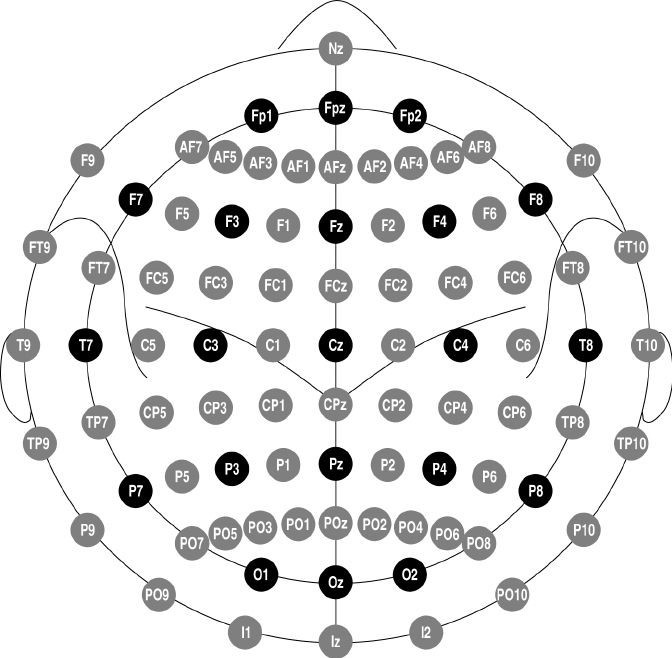
Extender el sistema 10-10 a un sistema 10-5 permite el uso de 300 o más puntos (320 han sido descritos) [7].
EEGLAB reconoce extensiones como .ced o .locs como archivos que contienen las ubicaciones de los canales, sin embargo, es posible importar estas desde una variedad de archivos (ver apartado Importación de chan-locs). La ubicación de los electrodos es útil para graficar scalp maps de las señales EEG, para estimar la localización de fuentes, entre otras aplicaciones, es por tanto que un dataset de EEGLAB debe contener la localización de los canales para su correcto análisis [10].
La subestructura EEG.chanlocs guarda la información acerca de la localización de los canales y sus nombres. Para acceder a la subestructura se escribe » EEG.chanlocs y devuelve las dimensiones de ésta y los tipos de datos que contiene; de la misma manera se puede escribir EEG.chanlocs(1) para acceder al primer canal y las coordenadas de éste [2] (ver figura 20).
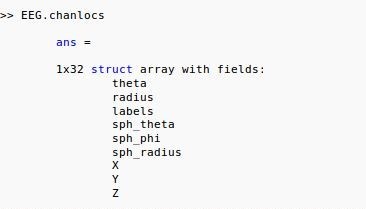
En el caso de la figura 20, la estructura es 1x32, lo cual indica un registro (señal) por cada uno de los 32 canales. En el campo labels se almacena el nombre del canal, mientras que en los demás campos hay información de su ubicación [2].
Para cargar o editar la localización de los canales mediante interfaz, ir al menú Edit > Channel locations. Una ventana preguntará el modelo craneal para la distribución de los electrodos, lea las indicaciones de la ventana, o simplemente presione Ok [8].
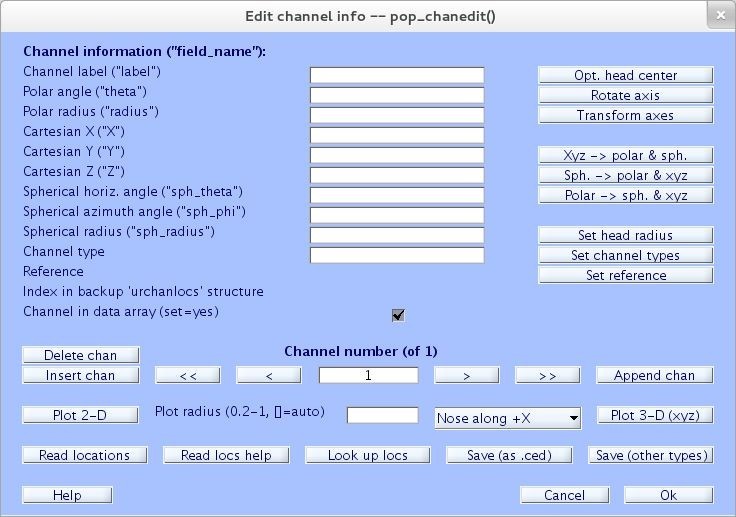
Puede ingresar la ubicación de los electrodos manualmente ingresando los valores respectivos en las casillas (no es necesario llenarlas todas, basta con ingresar la ubicación en coordenadas polares, esféricas o cartesianas), sin embargo, al modificar algún formato (cartesiano, polar o esférico) se debe actualizar al resto mediante los botones “Xyz -> polar & sph.”, “Sph. -> polar & xyz” y “Polar -> sph. & xyz”, pulse Ok para confirmar [11].
Si desea exportar la ubicación de los electrodos ingresada a un archivo, presione el botón Save (as .ced) para generar un archivo .ced, presione Save (other types) para generar otro formato de archivo.
El apartado Channel type es opcional e indica el tipo de canal (EEG, EMG, ECG,etc) [11].
Carga de chan-locs mediante archivo en EEGLAB:
Una vez en la ventana ilustrada en la figura 21, para cargar las localizaciones de los electrodos desde un archivo, pulse el botón Read Locations y seleccionar el archivo en cuestión [10].
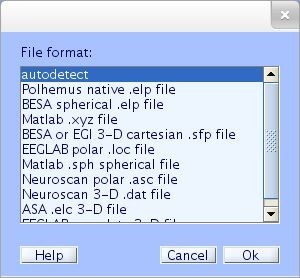
Al confirmar el archivo de ubicaciones, deberá indicarse el formato del archivo de posiciones usado en una ventana emergente como la visualizada en la figura 22. Simplemente presione Ok (autodetect), EEGLAB encontrará el formato usado basándose en la extensión del archivo [10].
Si desea cargar la localización de los canales desde un archivo usando código, ejecute la siguiente línea en la ventana de comandos de MATLAB.
>> EEG=pop_chanedit(EEG,'lookup',file_dir,'load',{ file_dir 'filetype' 'autodetect'});
Donde EEG representa la estructura asociada al dataset en cuestión y file_dir corresponde a la dirección del archivo chan-loc, por ejemplo:
>>file_dir='/home/Usuario/Documents/Chan-loc/Chan_locs_10-10_Standard-64-(BE
SA).ced';
Si desea abrir la ventana visualizada en la figura 21 mediante código, ejecute lo siguiente en la ventana de comandos de MATLAB:
>> pop_chanedit([]);
Ingresar ubicación de canales mediante labels estándar:
Los nombres (o labels) asociados a cada canal en distintos sistemas (como por ejemplo el 10-20 internacional y sus derivados: el 10% y el 5%) están estandarizados y corresponden a una locación única. EEGLAB contiene una base de datos con los labels de cada canal y su respectiva ubicación.
Si se quiere encontrar la posición de los electrodos de acuerdo a su label, una vez en la ventana mostrada en la figura 21 ingresar el nombre del electrodo en el campo Channel label (en caso de que el dataset no contenga los label de los canales) y pulsar el botón Look up locks. Al ingresar el label del canal tenga en cuenta que el nombre y el número (Channel number) del mismo coincidan, además de escribir correctamente la etiqueta (label) del electrodo.
Nota: los labels usados en el sistema 10-20 internacional y sus derivados son equivalentes, es decir, el electrodo C3 en el sistema 10-20 presenta la misma ubicación que su homónimo en el sistema 10% y en el 5%.
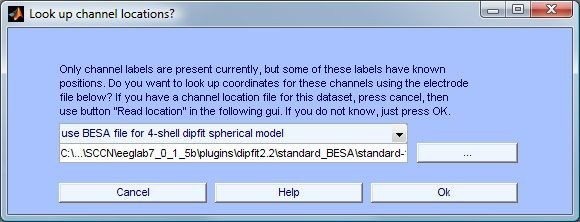
Nota: Si el dataset usado contiene las etiquetas asociadas a cada canal, al momento de ingresar al menú Edit > Channel locations, EEGLAB preguntará si se desea asignar las locaciones de los canales automáticamente basándose en su label, de acuerdo con el archivo “Standard-10-5-Cap385.sfp” localizado en “eeglabxxxx/function/resources” (figura 23) [11].
Importación de chan-locs:
En la tabla 2 se indican los formatos soportados por EEGLAB al momento de importar la locación de canales [11].
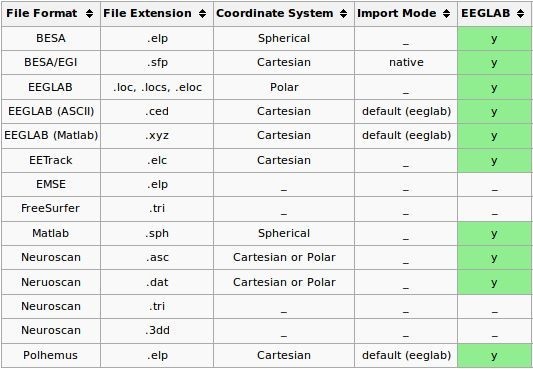
Los formatos marcados con la casilla Y (verde) en la columna EEGLAB pueden ser importados [11].
En las tablas 3, 4 y 5 se presenta el formato usado para registrar locaciones en extensión .loc (polares), .sph (esféricas) y .xyz (cartesianas) respectivamente. Hay que tener en cuenta que la primera fila es usada con propósitos ilustrativos y debe ser ignorada en un archivo real [11].
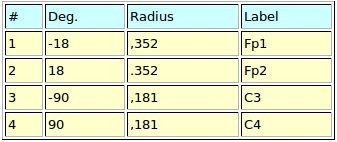
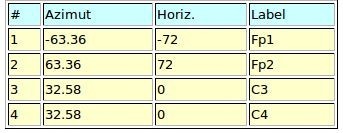
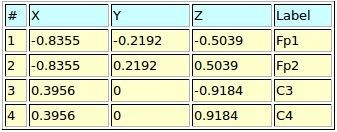
Visualización de electrodos:
Para asegurarse de que los electrodos fueron ubicados de manera correcta, se recomienda visualizar la posición de estos luego de la importación o carga. Es posible visualizar la posición de los electrodos desde la ventana pop_chanedit (figura 21) pulsando el botón Plot 2D (figura 24) o Plot 3D (xyz) (figura 25) [11].
Si se desea visualizar la ubicación de los electrodos desde el menú principal de EEGLAB, ir al menú Plot > Channel location > By name/number (ver figura 24). En el scalp map, al hacer clic sobre un electrodo en específico, cambiará la visualización entre número y nombre de éste [11].
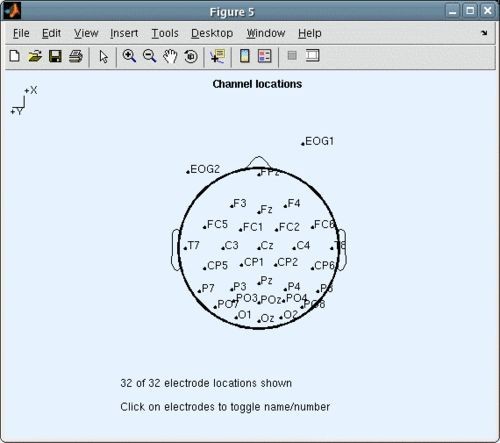
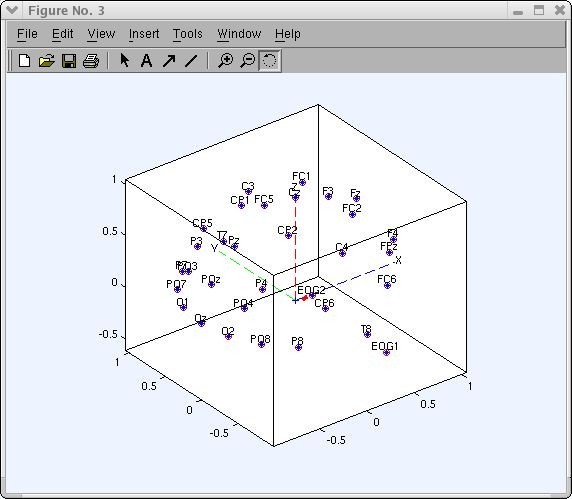
Por otro lado, si se desea visualizar la posición de los electrodos desde el script, se debe usar la función Topoplot. Ésta es usada por todo EEGLAB para graficar scalpmaps [10,11].
Extracción de épocas
Dividir el registro en secciones es una manera útil de extraer la información de interés para su posterior análisis. Al dividir el registro en partes o épocas es posible analizar la información obtenida en el registro de una manera organizada y práctica.La extracción de épocas en EEGLAB se hace con base en los eventos registrados.
EEG.epoch se encuentra ubicado dentro de la variable EEG, esta estructura contiene información acerca de las épocas asociadas al registro. Es una estructura similar a EEG.event, excepto que solo hay un registro para cada época, que contiene los siguientes campos: event, eventlatency, eventposition, eventtype, eventurevent. Los campos dentro de esta estructura son homólogos a los encontrados en EEG.events con excepción del campo event, el cual contien el índice de los eventos ocurridos en una época en específico. La longitud de la estructura hace referencia al número de épocas en el registro (ver figura 26) [2].

Para la extracción de épocas por medio de la interfaz gráfica se sigue de la siguiente manera: Tools > Extract Epochs [12].
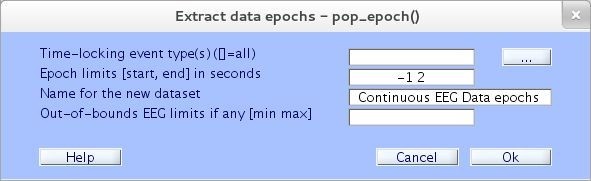
De acuerdo a la figura 27 se hace clic en el botón superior derecho “…” , y se presenta una ventana con los tipos de eventos disponibles (ver figura 28) [12].
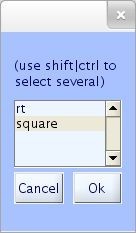
Se elige un tipo de evento, para el ejemplo de la figura 28 se escoge “square” y se presiona Ok.
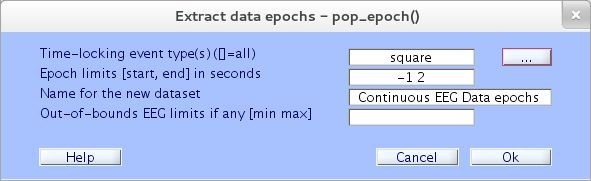
A continuación en la figura 29, en Epoch limits se ponen el tiempo en segundos del inicio y final de la época alrededor de donde ocurrió el evento en el experimento. Para este ejemplo se empieza en -1 (1 segundo antes del evento) y se termina en 2 (2 segundos luego del evento). También si se puede colocar un nombre descriptivo para el nuevo dataset y luego presionar Ok [12].
Seguidamente sale una ventana con opción para cambiar el nombre, guardar el dataset en una ubicación o aceptar lo que está por defecto, presionar Ok para confirmar (ver figura 30) [12].
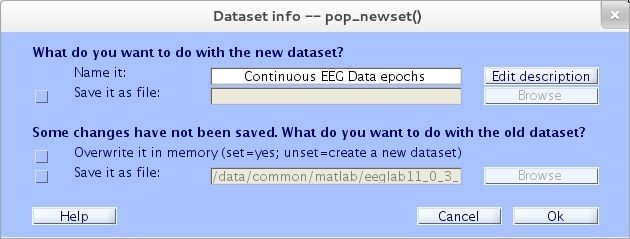
Para este caso, el estímulo tiene tres segundos de longitud. A menudo es mejor extraer épocas largas de los datos, para hacer posible la descomposición tiempo-frecuencia a bajas frecuencias (« 10 Hz) [12].
Extracción de los valores de la línea base:
La eliminación de la línea base de cada época es útil cuando tiene diferencias entre las épocas de los datos. Esto no es significativamente interpretable, pero sí puede generar sesgos en el análisis de los datos [11].
Después de haber extraído las épocas de los datos, la siguiente ventana (ver figura 31) saldrá automáticamente. También es posible remover la línea base, seleccionando en el menú Tools > Remove baseline [12].
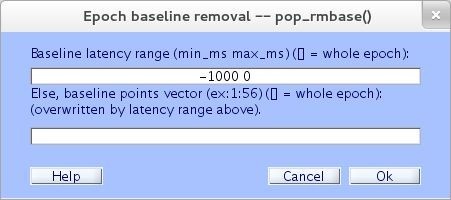
En la ventana de la figura 31 se puede especificar el periodo de la línea de base en milisegundos o en puntos de tiempo y también se puede establecer la ventana de latencia en cada época, a través de la cual se computa la media de la línea base para ser removida. El dataset original está por defecto sobrescrito por un dataset con una línea de base removida; además esto no es un método óptimo para seleccionar un periodo o valor de línea de base. Presione Ok para sustraer la línea de base o Cancel para no eliminarla [12].
Re-referenciar los datos:
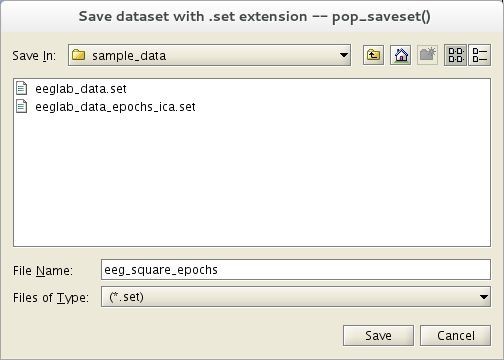
Luego de los pasos anteriores (extracción de épocas y eliminación de la línea de base) es buen momento para guardar el dataset bajo un nuevo nombre usando la ventana de pop_newset(), para guardar el actual dataset en cualquier otro momento, seleccione File > Save current dataset o File > Save current dataset as[12].
Aparece la ventana del buscador de archivos (figura 32), introduzca el nombre del dataset (que debe terminar con la extensión .set), presione Save y luego Ok para guardarlo con todos los datos incluidos hasta ese momento. Para la extracción de épocas por comandos de línea: el procedimiento anterior para la extracción de épocas por medio de código se realiza escribiendo en la ventana de comandos de MATLAB (o en un script) lo siguiente [5]:
>>EEG = pop_epoch( EEG, { 'square' }, [-1 2], 'epochinfo', 'yes');
No se debe olvidar primero remover la actividad de referencia o línea de base:
>> EEG = pop_rmbase( EEG, [-1000 0]);
Importación de épocas:
Importar información de épocas indica que éstas ya han sido extraídas de los datos continuos del EEG, y que el arreglo de MATLAB o el archivo de texto, contienen una entrada por época [4].
Importar épocas desde arreglo de MATLAB: Si se desea importar épocas, ir al menú File > Import epoch info > from Matlab array or ascii file; cabe resaltar que la importación de épocas involucra los mismos pasos que una importación de un dataset continuo,es decir, para importar las épocas de un dataset desde un arreglo en MATLAB, es necesario importar toda la señal, segmentada ya en épocas. Al importar un arreglo de MATLAB de 3 dimensiones, es automáticamente importado como un dataset dividido en épocas, siendo la primera dimensión interpretada como los canales del registro, la segunda como los puntos que contienen los datos, y la tercera como las épocas o intentos; para realizarlo seleccionar File > Import epoch info > from Matlab array or ascii file [4].
Importar épocas desde un archivo de texto (ascii): Para importar épocas desde un archivo de texto, ir al menú File > Import epoch info > from Matlab array or ascii file. En la tabla 6 se indica el formato necesario para el archivo [4].
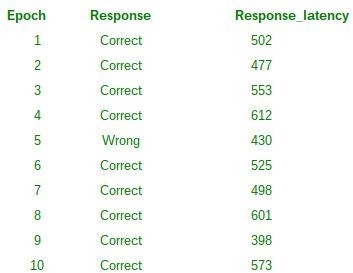
La figura 33 indica la ventana asociada a la importación de épocas mediante archivo de texto.
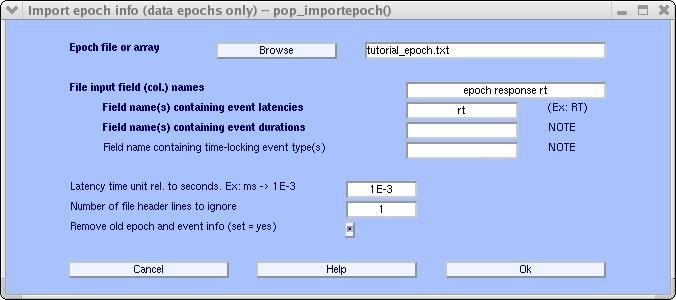
En el campo File input field (col.) names indique epoch response rt, siendo rt un acrónimo para el tiempo de reacción. En el campo Field name(s) containing latencies indique rt, este es el único campo que contiene información acerca de las latencias. En el campo Number of file header lines to ignore introduzca el número de líneas usadas en el encabezado del archivo, para indicar el nombre de las columnas. En el campo Latencies time unit rel. to seconds indique las unidades en las que se encuentran dados los tiempos de latencia, siendo 1E-3 equivalente a milisegundos. Active la casilla Remove old epoch and event info para sobreescribir los datos asociados a eventos y épocas [4].
Análisis de componentes independientes (ICA)
Imagine que está en una habitación, en la cual dos personas se encuentran hablando al tiempo. Usted cuenta con dos micrófonos ubicados en distintas posiciones dentro de la habitación. Los micrófonos reportan las señales X1(t) y X2(t), cada una de las cuales está conformada por la suma de ambas fuentes (persona 1 y persona 2) S1(t) y S2(t) en diferentes proporciones. Se tiene entonces que las señales X1(t) y X2(t) vienen dadas por las ecuaciones (1) y (2) [13]:
Donde los parámetros a nn dependen de factores como la distancia de los micrófonos a las personas y otros factores [13].
Es de interés conocer la forma de las señales S1(t) y S2(t), note que, si se conocen los parámetros a nn , es posible resolver las ecuaciones (1) y (2), encontrando los datos de interés [13].
Para conocer los parámetros a nn es útil asumir información acerca de las características estadísticas de las señales. Asumiendo que S1(t) es estadísticamente independiente de S2(t) en todo momento t, se obtienen resultados certeros al momento de encontrar las fuentes, teniendo en cuenta que en la práctica no es necesario que se cumpla esta condición a cabalidad [13].
Al igual que en el ejemplo anterior, las señales encontradas mediante EEG son una mezcla de señales producidas por fuentes (tanto neuronales como no neuronales) al momento de realizar el registro. Asumiendo que el proceso de mezcla (en el caso de EEG, debido al volúmen conductor) no agrega información nueva a las señales, además de que es lineal y pasivo, es posible, bajo ciertas consideraciones, estimar las fuentes de las señales registradas por los electrodos [13].
El análisis de componentes independientes (ICA por sus siglas en inglés) es un proceso de separación ciega de fuentes que permite encontrar las señales S1(t) y S2(t) (en el caso de EEG, fuentes neuronales o no neuronales) desde los datos obtenidos con X1(t) y X2(t) (señales adquiridas por los electrodos), asumiendo que las fuentes son estadísticamente independientes y que el número de fuentes es igual al número de sensores (micrófonos);esta última condición aplica para EEG[13].
Calcular componentes ICA mediante EEGLAB:
Para encontrar las componentes ICA mediante la interfaz gráfica de EEGLAB, ir al menú Tools > Run ICA. Se mostrará una ventana emergente como la mostrada en la figura 34 [14].
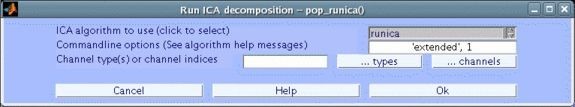
Es posible seleccionar canales de cierto tipo (por ejemplo que se incluyan canales EEG y EMG), o incluso una lista de canales para ejecutar el algoritmo ICA; mediante la opción Channel type(s) or channel indices se selecciona el tipo o grupo de canales a los cuales se les desea aplicar la descomposición. Hay que notar que EEGLAB admite editar el uso de distintos algoritmos de descomposición ICA y solo runica y jader son parte de la distribución estándar de EEGLAB, sin embargo, si se desea usar fastica instale la fastica toolbox, para luego incluirla en el path de MATLAB [14].
Las diferencias entre uno u otro algoritmo de descomposición no están del todo establecidas, sin embargo, el equipo de desarrollo de EEGLAB recomienda el uso de algoritmos Infomax ICA (runica/binica), debido a la estabilidad de las componentes encontradas, especialmente en su versión más rápida binica (para usar el algoritmo binica es necesario descargar el archivo binario en https://sccn.ucsd.edu/wiki/Binica, y luego modificar el archivo icadefs.m para que apunte al archivo binario correcto) [14].
Si se quiere ejecutar la descomposición ICA mediante código, hay que correr la siguiente línea en la ventana de comandos de MATLAB [14]:
>> EEG = pop_runica(EEG,'extended',1);
Los atributos ´extended´ y 1 son recomendados por el equipo de desarrollo de EEGLAB, con el fin de que el algoritmo detecte fuentes subgausianas.
Si se desea abrir la ventana mostrada en la figura 34 mediante código ejecute la siguiente línea en la ventana de comandos de MATLAB [14]:
>> pop_runica();
Graficar scalpmaps de componentes en 2D:
Para graficar los componentes ICA en 2D ir al menú Plot > Component maps > in 2-D, emergerá una ventana similar a la mostrada en la figura 35 [14].
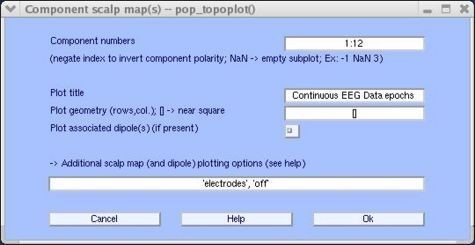
Luego de introducir el número de componentes a graficar, pulsar Ok. En la figura 36 se pueden observar los scalpmaps de algunas componentes ICA.
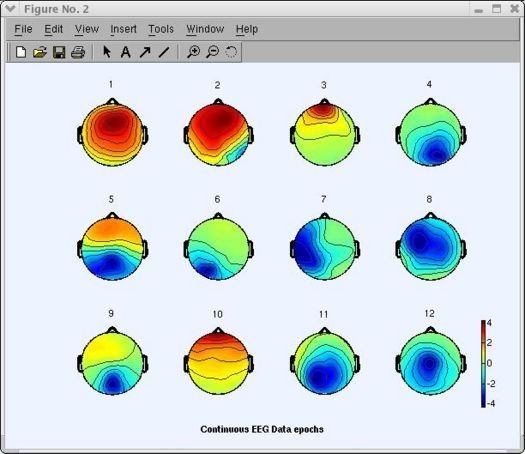
Nota: las unidades usadas para graficar las componentes (scalpmaps) son arbitrarias y las unidades usadas asignadas a la actividad ICA a lo largo del tiempo (scroll) también son arbitrarias. Sin embargo, la multiplicación de los valores observados en los escalpmaps y los datos ICA a lo largo del tiempo, tienen como resultado las mismas unidades de los datos originales (sin aplicar ICA) [14].
Graficar headplots de componentes en 3D:
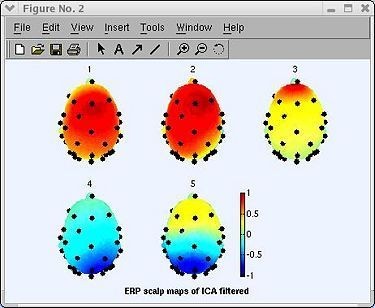
Para graficar la activación de los componentes en 3D ir al menú Plot > Component maps > in 2-D, emergerá una ventana como la mostrada en la figura 37 [14].
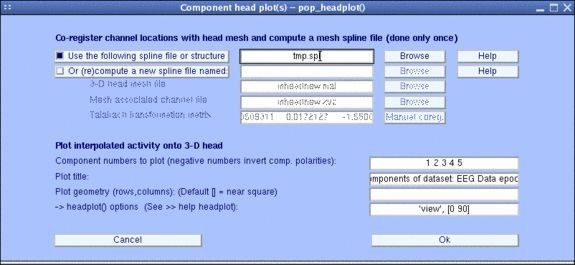
Seleccionar los componentes a graficar, junto con los electrodos a ubicar (si se desea) y presionar Ok. En la figura 38 se pueden observar los headplots de algunos componentes ICA.
Estudiando y removiendo componentes ICA:
Aprender a reconocer los tipos de componentes independientes puede requerir experiencia. Los principales criterios para determinar si un componente es 1 cognitivamente relacionado (neuronal), 2) artefacto muscular o 3) algún otro tipo de artefacto, son [14]:
- Scalpmap de la componente.
- Actividad de la componente en el tiempo (para ver, ir al menú: Plot > Component activation (scroll)).
- Espectro de potencia de la actividad del componente.
- La erpimagen.m de la componente.
Para estudiar las propiedades de los componentes y si se desea marcarlos para su rechazo (eliminación del aporte del componente a la señal), ir al menú tools > Reject data using ICA > Reject components by map y emergerá una ventana similar a la mostrada en la figura 39 [14].
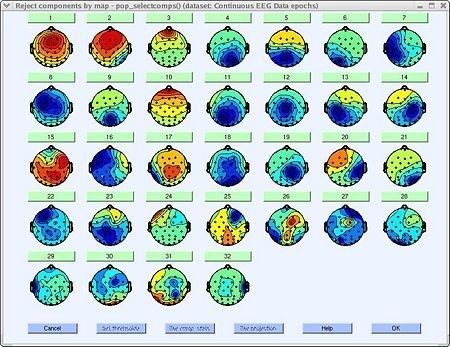
Seleccionando uno de los cuadros ubicados encima de los componentes, es posible visualizar las propiedades de estos [14].
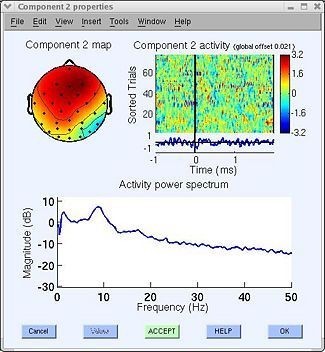
Características de componentes oculares: El componente 3 de la figura 39 muestra signos de estar asociado a un artefacto visual, estos artefactos suelen tener características como [14]:
● Decrecimiento suave de la potencia espectral (figura 40, inferior). ● El scalpmap muestra una proyección fuertemente frontal (figura 40, superior izquierda) ● La erpimágen evidencia movimientos oculares individuales (figura 40, superior derecha).
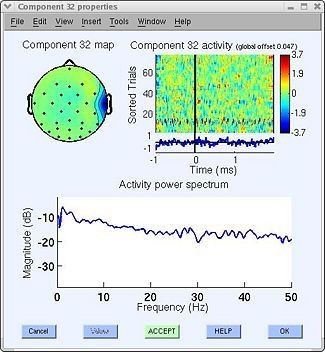
Características de componentes musculares: El componente 32 de la figura 39 muestra signos de estar asociado a un artefacto muscular, estos artefactos suelen tener características como [14]:
● Estar espacialmente localizados, activación temporal generalmente (figura 41, superior izquierda). ● Presentar alta potencia en frecuencias altas (20-50 Hz o más) (figura 41, inferior).
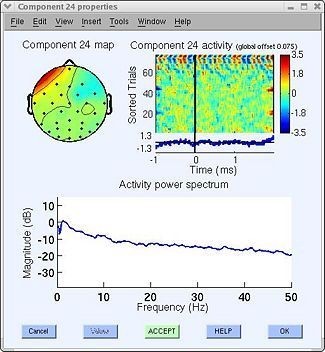
Características de artefactos asociados a electrodos sueltos: Un tipo usual de artefacto encontrado es el asociado a electrodos que, durante el registro, perdieron contacto o su impedancia asociada aumentó demasiado. En la figura 39 no está presente este tipo de artefacto (no con certeza), sin embargo, la componente 24 muestra signos de haber captado ruido de 60 Hz, especialmente luego del trial 65 (figura 42, superior derecha). Este tipo de artefactos suele presentar activación espacial localizada y no presentar representación de dipolo (como sí lo suelen hacer las fuentes neuronales) [14].
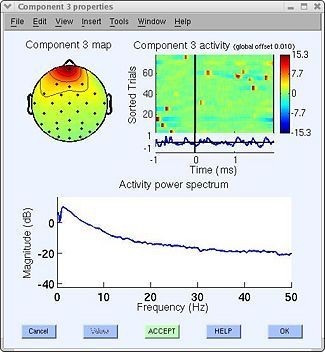
Características de fuentes posiblemente neuronales: El componente 2 de la figura 39 podría estar asociada a una fuente neuronal, este tipo de componentes pueden presentar características como [14]:
● Scalpmaps dipolares (figura 43, superior izquierda). ● Picos espectrales en frecuencias típicas de EEG,suelen presentarse picos a bajas frecuencias e ir descendiendo a medida que aumentan las frecuencias (figura 43, inferior). ● Activaciones a lo largo del tiempo asociadas a ERP (event related potentials), con lo cual quiere decir que el componente presenta un pico de actividad (positivo o negativo) luego del estímulo, no antes (figura 43, superior derecha).
Nota: si un componente parece ser mitad artefacto, mitad fuente neuronal, se sugiere dejarlo dentro del registro, o correr el algoritmo ICA nuevamente [14].
Rechazando (eliminando) componentes ICA de los datos:
Podría ser de utilidad eliminar la influencia de componentes asociados a artefactos musculares, visuales u otros en la señal EEG, sin embargo es importante tener en cuenta que, mediante este procedimiento no se está exento de eliminar posible información de utilidad.
Una vez seleccionado el componente deseado,emergerá una ventana similar a las vistas en las figuras 40, 41, 42 y 43, presionar el botón verde Accept y éste cambiará al estado de Reject (botón rojo). En este punto, el componente está marcado para su rechazo, sin embargo no está eliminado aún, para eliminar el componente ir al menú Tools > Remove components, seleccionar los componentes a eliminar y presionar Ok, emergerá una ventana como la mostrada en la figura 44. Si se seleccionó componentes a rechazar mediante el método Accept/Reject asociado al menú Tools > Reject data using ICA > Reject components by map, estas componentes aparecerán por defecto en el campo Component(s) to remove from data) [14].
Figura 44. ERP de los datos antes (azúl) y después (rojo) del rechazo de los componentes[14].
La ventana anterior mostrará los ERP de los datos antes (azúl) y después (rojo) del rechazo de los componentes, si se está de acuerdo con los resultados, presionar Yes, y en la ventana emergente ingresar los datos del nuevo dataset [14].
Si se intenta graficar los scaplmaps o headplots de los componentes ICA luego del rechazo, se encontrará que no se incluirán los eliminados. Si se desea, hay que ejecutar nuevamente los algoritmos ICA para obtener nuevas componentes [14].
Si se quiere encontrar menos componentes de los usualmente extraídos, se debe ingresar “pca”,”NN” en las opciones al momento de ejecutar los algoritmos, donde NN es el número de componentes a encontrar [14].
Referencias
[1] “Chapter 02: Writing EEGLAB Scripts - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_02:_Writing_EEGLAB_Scripts. [Consultado: 01- Sep- 2016].
[2] “A05: Data Structures - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/A05:_Data_Structures. [Consultado: 01- Sep- 2016].
[3] “Chapter 01: Loading Data in EEGLAB - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_01:_Loading_Data_in_EEGLAB.[Consultado: 02- Sep- 2016].
[4] “A01: Importing Continuous and Epoched Data - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/A01:_Importing_Continuous_Epoched_Data. [Consultado: 02- Sep- 2016].
[5] “Chapter 03: Event Processing - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_03:_Event_Processing. [Consultado: 01- Sep- 2016].
[6] “A02: Importing Event Epoch Info - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/A02:_Importing_Event_Epoch_Info. [Consultado: 01- Sep- 2016].
[7] V. Jurcak, D. Tsuzuki & I. Dan, “10/20, 10/10, and 10/5 systems revisited: Their validity as relative head-surface-based positioning systems”, NeuroImage, vol. 34, no. 4, pp. 1600-1611, 2007.
[8] “13. Electroencephalography”, Bem.fi, 2016. [En línea]. Disponible en: http://www.bem.fi/book/13/13.htm. [Consultado 09- Ago- 2016].
[9] R. Oostenveld and P. Praamstra, “The five percent electrode system for high-resolution EEG and ERP measurements”, Clinical Neurophysiology, vol. 112,no. 4, pp. 713-719, 2001.
[10] “Chapter 02: Channel Locations - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_02:_Channel_Locations. [Consultado: 01- Sep- 2016].
[11] “A03: Importing Channel Locations - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/A03:_Importing_Channel_Locations. [Consultado: 01- Sep- 2016].
[12] “Chapter 05: Extracting Data Epochs - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_05:_Extracting_Data_Epochs. [Consultado: 02- Sep- 2016].
[13] A. Hyvärinen & E. Oja, “Independent component analysis: algorithms and applications”, Neural Networks, vol. 13, no. 4-5, pp. 411-430, 2000.
[14] “Chapter 09: Decomposing Data Using ICA - SCCN”, Sccn.ucsd.edu, 2016. [En línea]. Disponible en: https://sccn.ucsd.edu/wiki/Chapter_09:_Decomposing_Data_Using_ICA. [Consultado: 01- Sep- 2016].